Un grupo de científicos ha desarrollado un programa informático fácil de usar que puede analizar rápidamente el ADN bacteriano de la infección de un paciente y predecir cómo actuarán los antibióticos y que error se producirá debido a la resistencia a los medicamentos. El software está siendo probado actualmente en tres hospitales del Reino Unido para ver si puede ayudar a acelerar el diagnóstico de las infecciones resistentes a los fármacos y permitir a los médicos orientar mejor la prescripción de antibióticos.
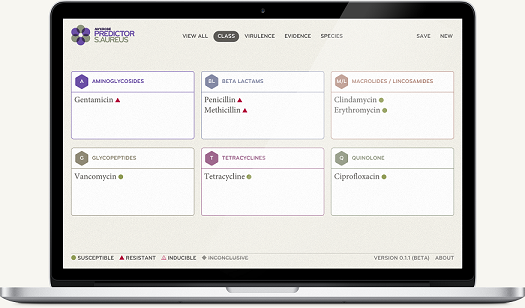
El software Mykrobe Predictor, desarrollado por el Dr. Zamin Iqbal y sus colegas del Centro Wellcome Trust de Genética Humana de la Universidad de Oxford, se ejecuta en un ordenador portátil estándar o tablet sin necesidad de ningún conocimiento especializado. El programa puede analizar todo el código genético de una bacteria en menos de 3 minutos, una vez que la muestra bacteriana ha sido cultivada y se ha secuenciado su ADN.
Un estudio de más de 4.500 muestras de pacientes retrospectivos, publicado hoy en la revista digital Nature Communications, muestra que el Mykrobe Predictor detecta con precisión la resistencia a los antibióticos en dos infecciones bacterianas potencialmente mortales: Staphylococcus aureus (que causa la infección por estafilococo resistente a la meticilina (MRSA) y la tuberculosis (TB).
El software está siendo probado en hospitales de Oxford, Brighton y Leeds como parte de un proyecto dirigido por el profesor Derrick Crook que, en colaboración con programas paralelos en la UCL y Cambridge University, tiene como objetivo desarrollar la secuenciación completa del genoma como una herramienta rutinaria para el diagnóstico y control de la infecciones. El programa está financiado por el Departamento de Salud de Reino Unido y el Wellcome Trust a través del Fondo conjunto Health Innovation Challenge.
Las infecciones resistentes a los medicamentos representan una grave amenaza para la salud mundial y podría significar que en el futuro las infecciones graves y mortales puedan volverse imposibles de tratar. El problema se desarrolla por selección natural. Al igual que otras especies, las bacterias están en constante evolución a través de sutiles cambios en su ADN. Algunos de estos cambios los hacen resistentes a ciertos medicamentos, lo que significa que tienen más probabilidades de sobrevivir y transmitir sus rasgos de resistencia a otras bacterias.
Una de las mejores maneras de prevenir la propagación de bacterias resistentes es asegurarse de que los pacientes con una infección son tratados rápidamente con el tipo correcto de antibióticos. Pero para saber qué cepa de bacteria en concreto está causando la infección de un paciente y a qué medicamentos es resistente, los médicos deben llevar a cabo pruebas de sensibilidad a los medicamentos, donde se aplican diferentes antibióticos a la bacteria en una placa de Petri para ver si la matan. Este proceso puede tardar días o incluso meses cuando se trata de infecciones de crecimiento lento como la tuberculosis.
Los científicos creen que podrían obtener la misma información mucho más rápido buscando directamente las mutaciones que se sabe que causan resistencia en la secuencia de ADN de la bacteria. Sin embargo, la interpretación de la información genética a menudo requiere una gran cantidad de potencia de cálculo y la experiencia de especialistas bioinformáticos.
El Mykrobe Predictor agiliza este proceso automatizando el análisis del genoma, cotejando la secuencia del ADN de la bacteria con cepas anteriores para buscar mutaciones causantes de la resistencia y presentando información sobre el virus en un formato fácil de entender.
En este estudio, el software fue capaz de detectar la resistencia a los cinco antibióticos de primera línea en más del 99% de los casos de Staphylococcus aureus, igualando el rendimiento de las pruebas de sensibilidad a los fármacos tradicionales. Para la tuberculosis, donde la base genética de la resistencia a los medicamentos es menos conocida, el Mykrobe Predictor también se igualó a las pruebas de ADN vigentes (que observan fragmentos de DNA, pero la secuencia completa), al detectar un 82,6% de las infecciones resistentes, alrededor de unas 5-16 semanas más rápido que las pruebas tradicionales de sensibilidad a los medicamentos. Sin embargo, a diferencia de las pruebas estándar de ADN ‘basadas en fragmentos’, el Mykrobe Predictor se puede actualizar rápidamente con una simple actualización de software que permite a los investigadores detectar nuevas mutaciones de resistencia a medida que evolucionan.
Una ventaja más del Mykrobe es que puede identificar las infecciones en cuerpos de pacientes que contienen una combinación de bacterias resistentes a los fármacos y sensibles a los medicamentos. En este estudio, la capacidad de distinguir entre estas sub-poblaciones bacterianas dio al Mykrobe una ventaja sobre las pruebas convencionales respecto a la detección de la resistencia a los fármacos antituberculosos de segunda línea. Esto es importante para el diagnóstico de infecciones tales como la “Tuberculosis extensivamente resistente a los medicamentos” (XDR-TB), que es resistente a, al menos, cuatro de los medicamentos básicos de la TB y es considerada por la Organización Mundial de la Salud como una amenaza global para la salud pública.
El autor principal del artículo, el Dr. Zamin Iqbal, del Centro <i<>Wellcome Trust de Genética Humana de la Universidad de Oxford, comentó:
“Una de las barreras para hacer todo el genoma secuenciar una parte rutinaria de la atención NHS es la necesidad de los ordenadores y la experiencia de gran alcance para interpretar las masas de datos complejos. Nuestro software gestiona los datos de forma rápida y presenta los resultados a los médicos y enfermeras en formas que son fáciles de entender, y así puedan utilizarlos instintivamente para tomar mejores decisiones respecto al tratamiento“.
El co-autor Derrick Crook, profesor, microbiólogo consultor en el Hospital John Radcliffe y Director del Servicio Nacional de Infecciones de Inglaterra, dijo:
“La secuenciación del genoma tiene el potencial de transformar la forma de diagnosticar y tratar las infecciones bacterianas, pero uno de los principales retos es el desarrollo de las herramientas adecuadas para que podamos extraer esta información de forma rápida y asequible. Nuestro objetivo final es ser capaces de proporcionar información completa de un patógeno dentro de las 24 horas de cultivo, vinculando esta información a una base de datos nacional de vigilancia para establecer un tratamiento más oportuno y más específico para el paciente“.
El Dr. Stephen Caddick, Director de Innovaciones en el Wellcome Trust, añadió:
“Las infecciones resistentes a los medicamentos representan una grave amenaza para la salud pública mundial. Los antibióticos que antes eran salvavidas están en peligro de convertirse en inútiles, y con el tiempo podríamos ver cómo infecciones menores vuelven a convertirse en un importante problema de salud pública. Necesitamos urgentemente nuevas estrategias de diagnóstico que nos permitan hacer un mejor uso de los antibióticos, y con ello salvaguardar la eficacia de los antibióticos existentes y los nuevos medicamentos que se desarrollen en el futuro“.
..Susana Calvo