Redacción
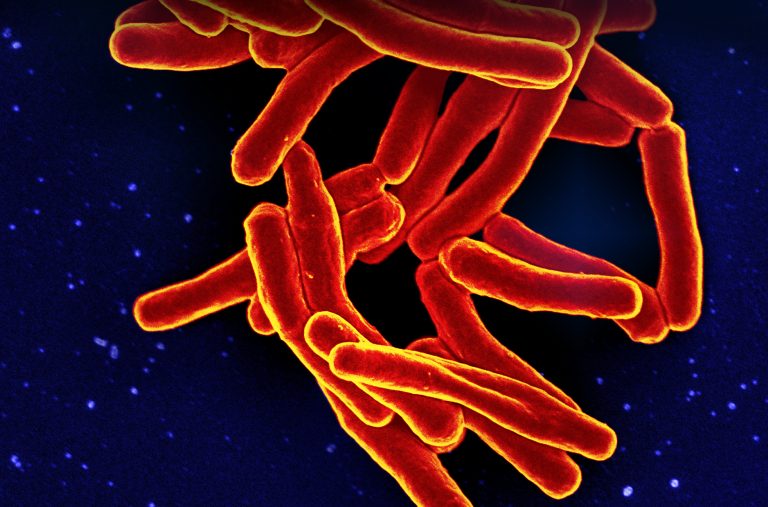
Entre las diez bacterias que causan más muertes por su resistencia a antibióticos está Mycobacterium tuberculosis, causante de la tuberculosis, la enfermedad infecciosa más letal en el planeta hasta la aparición del Covid-19. Disponer de diagnósticos que detecten de forma rápida y precisa las resistencias a antibióticos es vital para personalizar el tratamiento y evitar el desarrollo de resistencias adicionales. Por ello, un equipo de investigación del Instituto de Biomedicina de Valencia (IBV), del Consejo Superior de Investigaciones Científicas (CSIC), junto con centros hospitalarios de la Comunidad Valenciana, ha realizado un estudio que aplica técnicas de secuenciación masiva a 785 muestras de pacientes obtenidas en 25 hospitales para predecir posibles resistencias a fármacos de la bacteria que produce la tuberculosis.
Los resultados, publicados en la revista The Lancet Microbe, hallaron 13 nuevos casos de tuberculosis resistente frente a los encontrados con el diagnóstico de rutina, que utiliza el método de referencia actual, basado en el cultivo de muestras. Esto abre la puerta a utilizar la secuenciación masiva del ADN como método de referencia, acortando los plazos de detección de resistencias.
Los investigadores hallaron 13 nuevos casos de tuberculosis resistente frente a los encontrados con el diagnóstico de rutina
En la actualidad, los hospitales utilizan un método basado en el cultivo de muestras del paciente infectado y, en el caso de Mycobacterium tuberculosis, tardan semanas para obtener el fenotipo de la bacteria. Estas resistencias están causadas por mutaciones genéticas, de forma que cambios específicos en el ADN se asocian siempre con resistencia a antibióticos concretos.
Ahora, el equipo investigador ha utilizado técnicas de secuenciación masiva del material genético de esta bacteria para predecir a qué antibióticos es resistente y cómo esa predicción podría mejorar el diagnóstico y tratamiento de la enfermedad. De esta forma, se pueden determinar, con rapidez y precisión, la secuencia de ADN de cualquier ser vivo, y su principal ventaja es que pueden ser usadas a gran escala.
Nuevos casos de tuberculosis resistente
En concreto, el proyecto del IBV consiste en evaluar la capacidad de predicción de resistencias utilizando el catálogo de la OMS, particularmente en un entorno de bajas resistencias como es la Comunidad Valenciana. “Para ello secuenciamos el ADN extraído de muestras de pacientes con tuberculosis y obtenemos por predicción genómica el perfil de resistencia de los fármacos usados en el tratamiento”, explica Victoria Furió, investigadora en Biología Evolutiva y Microbiología del Instituto de Biomedicina de Valencia.
Victoria Furió: “Secuenciamos el ADN extraído de muestras de pacientes con tuberculosis y obtenemos por predicción genómica el perfil de resistencia de los fármacos usados en el tratamiento”
Después, añade que “comparamos el perfil de resistencias obtenido con secuenciación masiva con el obtenido mediante testado fenotípico, que es el método de referencia actual, y comprobamos cuán precisa es la predicción y la mejora que puede representar en el diagnóstico”. Los investigadores estudiaron los cuatro antibióticos de primera línea de tratamiento de la tuberculosis, obteniendo una especificidad superior al 99,5% y un rango de sensibilidad entre el 85% y el 50% en función del antibiótico mediante la secuenciación masiva del ADN.
“Encontramos una gran complementariedad entre los dos métodos. Si bien la genómica predice correctamente la gran mayoría de resistencias, hubo ejemplos solo detectados por el cultivo. También encontramos ejemplos en la otra dirección, donde la genómica superaba al método estándar. De hecho, gracias a la predicción genómica pudimos detectar dos nuevos casos de tuberculosis multidrogorresistente y once casos de tuberculosis monorresistente a fluoroquinolonas”, señala el investigador del CSIC, Iñaki Comas.
La secuenciación masiva en el diagnóstico de resistencias permite un gran número de aplicaciones gracias al acceso al genoma completo de la bacteria
Entre las principales ventajas de la secuenciación masiva en el diagnóstico de resistencias está la rapidez, “si se realiza en el momento que se detecta un cultivo positivo para tuberculosis”, apunta Comas. Otra ventaja es el gran número de aplicaciones que proporciona el acceso al genoma completo de la bacteria.
Esto permite, simultáneamente, establecer el perfil de resistencias para todo el espectro de fármacos (no sólo los cuatro principales); identificar casos de resistencias a antibióticos que se pierden por el método de referencia; y obtener información para realizar análisis epidemiológicos y de transmisión. “Con una sola determinación del genoma podemos predecir las resistencias y las relaciones epidemiológicas entre casos”, concluye Comas.