Redacción
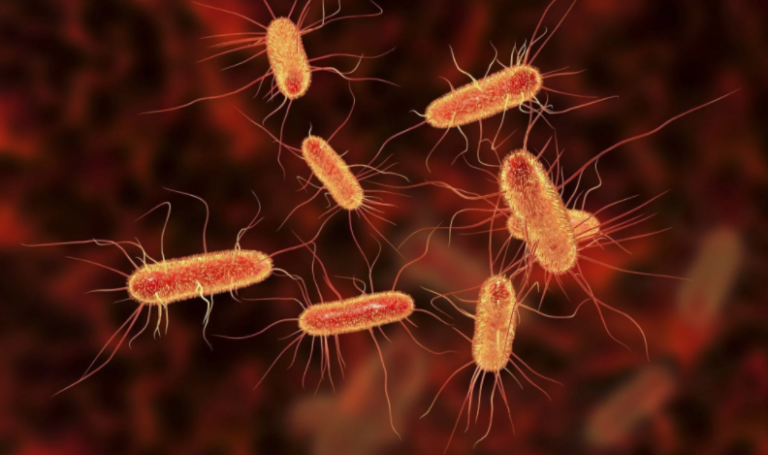
Científicos del Ciber forman parte del primer estudio multicéntrico en el que se ha caracterizado mediante secuenciación del genoma completo un elevado número de aislados de Escherichia coli, causantes de infecciones bacterianas graves.
Las características de virulencia y resistencia a antimicrobianos de los aislados de Escherichia coli de estos pacientes no se distribuyen aleatoriamente entre los distintos clones
El estudio Probac-EC demuestra que la estructura poblacional de la bacteria en pacientes con infección grave es altamente compleja y que las características de virulencia y resistencia a antimicrobianos de estos microorganismos no se distribuyen aleatoriamente entre los distintos clones. Sus resultados se han publicado en la revista Lancet Microbe.

La investigación, realizada en 22 hospitales de España bajo la coordinación del Hospital Virgen Macarena, proporciona una caracterización genética exhaustiva de los aislados de Escherichia coli en pacientes que presentaban una respuesta desregulada a la infección, es decir, sepsis o shock séptico.
Se han analizado los Escherichia coli de 224 pacientes procedentes de 22 hospitales españoles
Esta caracterización servirá como base para estudios adicionales que permitan saber cuál es la contribución de los factores microbiológicos de esta bacteria a la presentación clínica grave. Los investigadores también esperan que este conocimiento tenga un potencial uso para el desarrollo de intervenciones diagnósticas, preventivas o terapéuticas.
Para este estudio se han analizado los Escherichia coli de 224 pacientes, reclutados para la cohorte Probac procedentes de 22 de los 26 hospitales españoles. El estudio ha desvelado una amplia diversidad clonal en los aislados, aunque cuatro de ellos causaron casi la mitad de los casos. Los aislados de estos clones predominantes mostraron, en general, un mayor número de factores de virulencia, y una asociación variable con resistencia a antibióticos.
El estudio ha desvelado una amplia diversidad clonal en los aislados aunque predominan cuatro, que mostraron mayor número de factores de virulencia
La investigación forma parte del desarrollo de la tesis doctoral de la Investigadora del Servicio de Enfermedades Infecciosas y Microbiología del Hospital Universitario Virgen Macarena, Natalia Maldonado y cuenta con la financiación del Instituto de Salud Carlos III. Además ha recibido soporte del Centro de Investigación Biomédica en la Red Enfermedades Infecciosas (Ciberinfec), el Instituto de Biomedicina de Sevilla (IBiS), la Fundación para la Gestión de la Investigación en Salud de Sevilla (Fisevi) y la Universidad de Sevilla.