..Redacción.
Un equipo de investigadores del Instituto de Biomedicina de Valencia del Consejo Superior de Investigaciones Científicas (IBV-CSIC) ha identificado nuevos genes responsables de la resistencia a isoniazida, el principal tratamiento de la tuberculosis. Para ello, realizaron un estudio a gran escala de los mecanismos de resistencia de Mycobacterium tuberculosis, la bacteria que causa la tuberculosis, a la isoniazida.
Mediante el uso de técnicas novedosas de genómica funcional, el equipo del IBV-CSIC analizó todos los genes de la bacteria para determinar los implicados en el mecanismo de resistencia al fármaco. Después, se compararon los resultados con datos clínicos. Esta técnica permitirá evaluar nuevos fármacos contra la enfermedad y anticiparse a posibles resistencias.
El equipo del IBV-CSIC analizó todos los genes de la bacteria Mycobacterium tuberculosis para determinar los implicados en el mecanismo de resistencia a soniazida
 Los resultados del estudio, publicados en Communications Biology, han permitido encontrar nuevos genes que, hasta ahora, no se habían relacionado con resistencia a isoniazida. Junto a la rifampicina, son los dos antibióticos más importantes en el tratamiento de la tuberculosis. La resistencia a estos dos antibióticos aumenta las probabilidades de fallo del tratamiento en más del 40% de los pacientes. En cambio, si no hay resistencias, las probabilidades de fallo son de menos del 5%. A día de hoy son dos antibióticos clave para los que no existe una alternativa viable.
Los resultados del estudio, publicados en Communications Biology, han permitido encontrar nuevos genes que, hasta ahora, no se habían relacionado con resistencia a isoniazida. Junto a la rifampicina, son los dos antibióticos más importantes en el tratamiento de la tuberculosis. La resistencia a estos dos antibióticos aumenta las probabilidades de fallo del tratamiento en más del 40% de los pacientes. En cambio, si no hay resistencias, las probabilidades de fallo son de menos del 5%. A día de hoy son dos antibióticos clave para los que no existe una alternativa viable.
Aunque la isoniazida lleva décadas en uso, aún existen interrogantes sobre cómo la bacteria adquiere resistencia. Aproximadamente en un 15% de los aislados clínicos se desconoce la mutación o mutaciones causantes de la resistencia.
Victoria Furió: “Hemos usado una aproximación basada en la genómica funcional, que consiste en determinar de forma experimental la función de todos los genes del genoma”
“En este estudio buscamos genes implicados en la resistencia a isoniazida que no hayan sido descritos con anterioridad. Hemos usado una aproximación basada en la genómica funcional, que consiste en determinar de forma experimental la función de todos los genes del genoma”, explica Victoria Furió, investigadora de la Unidad Genómica de la Tuberculosis del IBV.
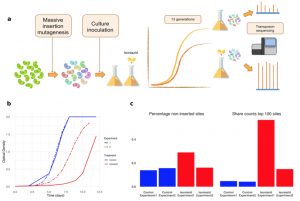
Secuenciación de ADN con transposones
Los autores utilizaron la secuenciación mediada por transposón. Los transposones son secuencias de material genético (ADN) con capacidad de ‘saltar’ dentro del genoma o incluso entre genes. Cuando un transposón se inserta en medio de un gen impide que se pueda expresar correctamente y ejercer su función. “Si una cepa de Mycobacterium tuberculosis con esta mutación crece muy bien en presencia del antibiótico isoniazida sabemos que ese gen en particular está asociado con resistencia a ese antibiótico”, aclara Furió.
Uno de los grandes beneficios de este trabajo es que “el conjunto de técnicas desarrolladas es aplicable a cualquier otro antibiótico”, asegura la investigadora
Este proceso lo repitieron con todos los genes de la bacteria. Para ello, generaron un conjunto de mutantes del bacilo de la tuberculosis, cada uno con una única inserción en un gen diferente. Después, monitorizaron su crecimiento usando la secuenciación mediada por transposón. “Así hemos podido ver que algunos mutantes crecen extremadamente bien en presencia de isoniazida mientras que otros lo hacen muy mal; obteniendo una lista de genes asociados a resistencia. Finalmente, hemos validado nuestros resultados usando secuencias de cepas clínicas de Mycobacterium tuberculosis”, detalla Furió.
Los resultados de este estudio se han validado utilizando una colección global de cepas de tuberculosis. Gracias a una gran cantidad de datos sobre mutaciones en la bacteria, los investigadores consiguieron relacionar los genes obtenidos en este trabajo con datos clínicos. Uno de los grandes beneficios de este trabajo es que “el conjunto de técnicas desarrolladas es aplicable a cualquier otro antibiótico”, asegura la investigadora. Esto es especialmente relevante para la evaluación de nuevos fármacos contra la tuberculosis y para anticiparse a posibles mecanismos de resistencia.
Noticias complementarias
- Expertos en tuberculosis auguran una sexta ola de Covid-19 en España y piden mantener el control de otras enfermedades infecciosas
- Las muertes por tuberculosis aumentan por primera vez en más de una década como consecuencia de la pandemia
- La nueva vacuna contra la tuberculosis diseñada por la Universidad de Zaragoza protege mejor que la actual vacuna BCG